Mit der Weiterentwicklung (stamm-)zellbasierter Therapien rückt zunehmend die Frage in den Vordergrund, ob diese Produkte in der Anwendung am Menschen sicher sind. Ein potentiell tumorigenes Risiko durch genetisch instabile oder veränderte Zellen sollte im Vorfeld der Zelltherapie analysiert werden. Vergleichende (molekular) zytogenetische Untersuchungstechniken werden als geeignet betrachtet, um zur Sicherheit in der Zelltherapie beizutragen. Grundsätzlich steht die Frage der genetischen Integrität der zu verwendeten Zellen im Vordergrund.
Unsere Expertise
Die Forschungsgruppe Authentifzierung, Stabilität und Identität von Zellen (ASIC) verfügt über eine langjährige genetische Expertise und bietet parallel zu ihren neuroonkologischen Forschungsaktivitäten nachfolgend aufgeführte Leistungen seit 2007 an. Neben der kontinuierlichen Unterstützung von Forschungsprojekten war die Arbeitsgruppe auch involviert in Studien zur Tumorigenität in der Zelltherapie und der Entwicklung regulatorischer Prozesse mit der European Medicine Agency (reflection paper on stem-cell based medical products, 2011; Risk of tumorigenicity in mesenchymal stromal cell based therapies--Bridging scientific observations and regulatory viewpoints, Cytotherapy 2013).
Es wird internen und externen Forschungseinrichtungen die Authentifizierung von Zelllinien wie auch (Stamm-) zellen mittels klassischer Analysetechniken und Mikroarray-Technologien angeboten. Häufig sind in diesem Zusammenhang folgende Fragen zu klären:
- Stimmt die Identität der gewünschten Zelllinie?
- Hat diese Zelllinie im Laufe der Kultivierung chromosomale Veränderungen akkumuliert?
- Sind die (Stamm)zellen für therapeutische Konzepte frei von sichtbaren Zeichen chromosomaler Instabilitäten?
Umfassende und vergleichende Analysen zur Prüfung der Zell- Authentizität oder genetischen Integrität tragen dazu bei, mangelhafte oder sogar falsche Forschungsergebnisse zu verhindern und unterstützen eine effizientere Verwendung von Ressourcen im Rahmen konzeptioneller Fragestellungen wie auch in der (prä-) klinischen Forschung.
Untersuchungsmethoden
Unsere Untersuchungsmethoden basieren auf zytogenetischen und molekularzytogenetischen Techniken:
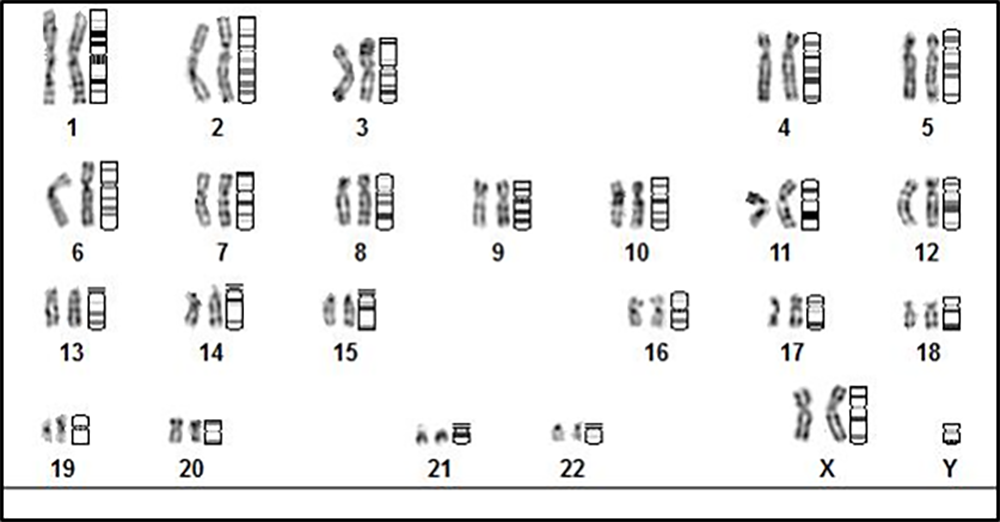
Die GTG-Technik ist eine Trypsin-induzierte Giemsa-Färbung, mit der ein reproduzierbares Bandenmuster aufgrund der Basenzusammensetzung, der Chromatinformation, der Gendichte, der repetitiven Sequenzen und des Replikationszeitpunktes erzeugt wird. Der hiermit ermittelbare Karyotyp erfasst die Gesamtheit der charakteristischen numerischen und strukturellen Informationen des Chromosomenbestandes auf Einzelzellbasis. Die Nachweisgrenze chromosomaler Aberrationen liegt bei ≥ 5Mb.
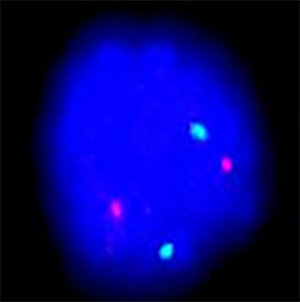
Die Fluoreszenz in situ Hybridisierung (FISH) ist eine Technik bei der chemisch markierte DNA-Sonden direkt auf Metaphasen / Interphasezellen hybridisiert und durch Fluoreszenzsignale sichtbar gemacht werden (z.B. DNA-Sonden, die durch modifizierte Nukleotide mit Reportermolekülen, wie Biotin gekennzeichnet sind). Mit dieser Methode sind, in Abhängigkeit von der Sondengröße, kleinere chromosomale Veränderungen ab 60Kb nachweisbar. Grundsätzlich können jeweils unterschiedliche chromosomale Regionen dargestellt werden (locusspezifische Regionen, Zentromerregionen, Subtelomer- Regionen, komplette Chromosomen).
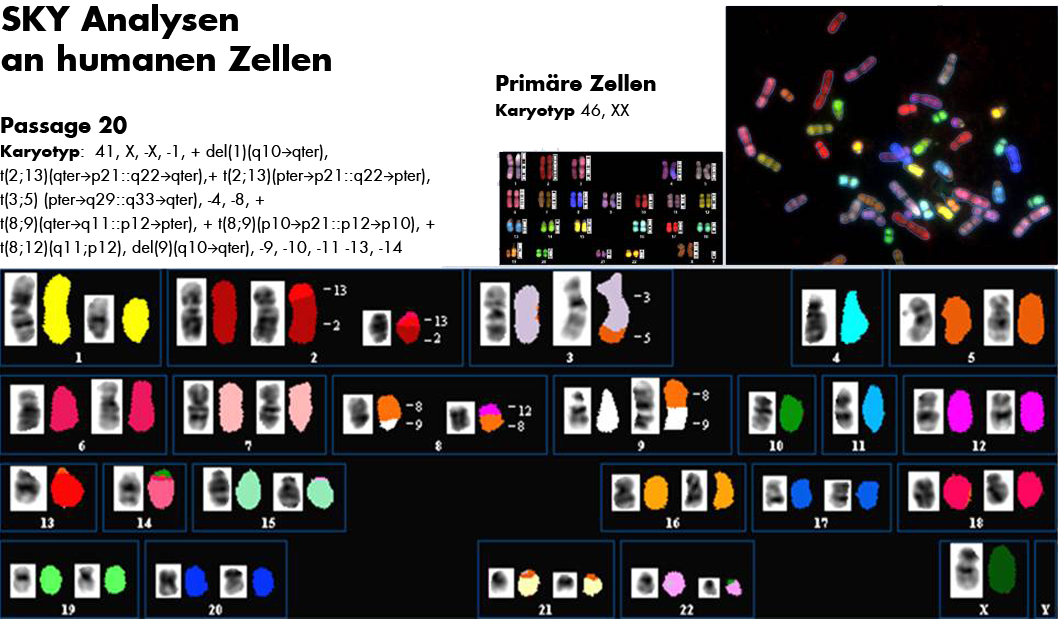
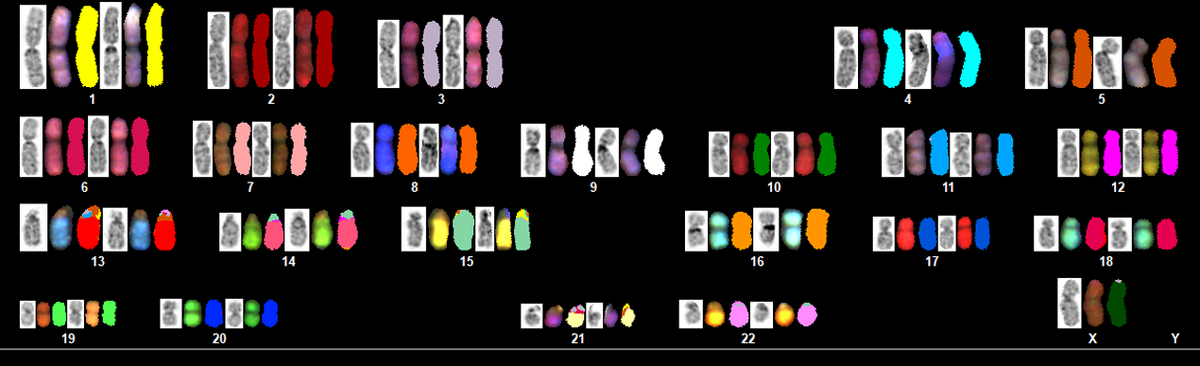
Die Spektrale Karyotypisierung (SKY-Technik) ist eine Vielfarbentechniken unter Involvierung von 24 chromosomenspezifischen DNA- Sonden mit Markierung durch kombinierte Fluorochrome. Mit dieser Technik ist ein Überblick über alle chromosomalen Regionen möglich, sodass die Erkennung von kryptischen balancierten/unbalancierten Translokationen und komplexen Rearrangements ab einer Größenordnung von 2-3 Mb möglich ist. Des Weiteren eignen sich diese Methoden zur Identifizierung von zusätzlichen und/oder konventionell nicht zuordenbaren chromosomalen Material (z.B. Markerchromosomen).
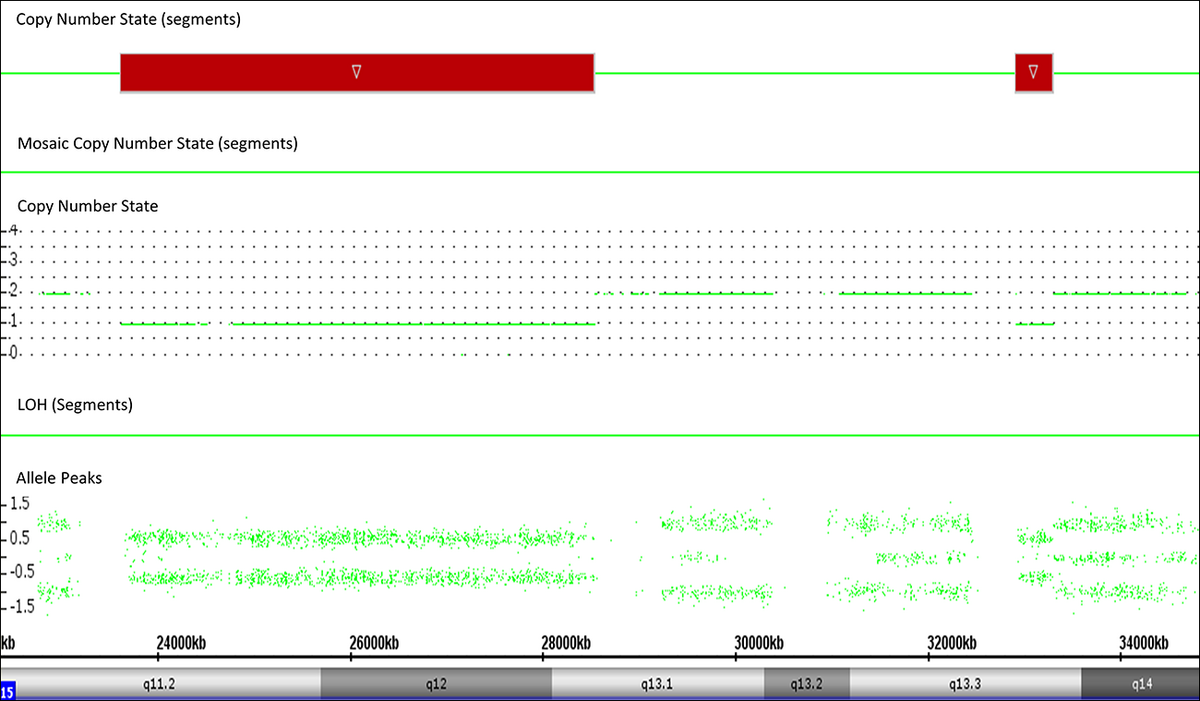
Die SNP-array Karyotypisierung ist eine Methode zur genomweiten Identifizierung allelischer Imbalancen mittels hochauflösenden Nachweises von Einzelbasen- Polymorphismen mit einem Auflösungsvermögen von etwa 10kb (in Abhängigkeit des verwendeten Arrays). Mit dieser Technik kann daher die Bestimmung der Kopienzahl und der Verlust an Heterozygotie parallel für das gesamte Genom in einer hohen Auflösung durchgeführt werden. Diese hochauflösende Technologie erlaubt die Identifizierung kleinerer genomischer Aberrationen.
nur auf Anfrage
Referenzen/Anwendungsbeispiele
Nachstehend finden Sie eine Auswahl unserer Referenzen:
- SNP array genomic analysis of matched pairs of brain and liver metastases in primary colorectal cancer
Vivian‑Pascal Brandt, Heidrun Holland, Marco Wallenborn, Ronald Koschny, Clara Frydrychowicz, Mandy Richter, Lydia Holland, Ulf Nestler & Caroline Sander
Journal of Cancer Research and Clinical Oncology, 149:18173–18183 (2023)
doi: 10.1007/s00432-023-05505-4 - Efficient high-precision homology-directed repair-dependent genome editing by HDRobust
Stephan Riesenberg, Philipp Kanis, Dominik Macak, Damian Wollny, Dorothee Düsterhöft, Johannes Kowalewski, Nelly Helmbrecht, Tomislav Maricic & Svante Pääbo
Nature Methods volume 20, 1388–1399 (2023)
doi: 10.1038/s41592-023-01949-1 - Stirred suspension bioreactors maintain naive pluripotency of human pluripotent stem cells
Leili Rohani, Breanna S. Borys, Golsa Razian, Pooyan Naghsh, Shiying Liu, Adiv A. Johnson, Pranav Machiraju, Heidrun Holland, Ian A. Lewis, Ryan A. Groves, Derek Toms, Paul M. K. Gordon, Joyce W. Li, Tania So, Tiffany Dang, Michael S. Kallos & Derrick E. Rancourt
Communications Biology volume 3, Article number: 492 (2020)
doi: 10.1038/s42003-020-01218-3 -
Molecular profiling analyses of glioblastoma stem-like cells and glioblastoma tissue
Marco Wallenborn, Li-Xin Xu, Holger Kirsten, Leili Rohani, Daniela Rudolf, Peter Ahnert, Christian Schmidt, Ronny M. Schulz, Mandy Richter, Wolfgang Krupp, Wolf Mueller, Adiv A. Johnson, Jürgen Meixensberger & Heidrun Holland
PLoS One, 15(7), (2020)
doi: 10.1371/journal.pone.0234986 - Simultaneous precise editing of multiple genes in human cells
Stephan Riesenberg, Manjusha Chintalapati, Dominik Macak, Philipp Kanis, Tomislav Maricic & Svante Pääbo
Nucleic Acids Research, Vol. 47, No. 19 (2019)
doi: 10.1093/nar/gkz669 - Altered neuronal migratory trajectories in human cerebral organoids derived from individuals with neuronal heterotopia
Johannes Klaus, Sabina Kanton, Christina Kyrousi, Ane Cristina Ayo-Martin, Rossella Di Giaimo, Stephan Riesenberg, Adam C. O’Neill, J. Gray Camp, Chiara Tocco, Malgorzata Santel, Ejona Rusha, Micha Drukker, Mariana Schroeder, Magdalena Götz, Stephen P. Robertson, Barbara Treutlein & Silvia Cappello
Nature Medicine 25, 561–568 (2019)
doi 10.1038/s41591-019-0371-0, 2019 - Molecular Cytogenetics and Quality Control: Clinical Guardians for Pluripotent Stem Cells
Leili Rohani, Adiv A. Johnson, Pooyan Naghsh, Derrick E. Rancourt, Henning Ulrich & Heidrun Holland
Stem Cells Translational Medicine, Volume 7, Issue 12, 867–875 (2018)
doi: 10.1002/sctm.18-0087 - Comprehensive high-resolution genomic profiling and cytogenetics of human chondrocyte cultures by GTG-banding, locus-specific FISH, SKY and SNP array
Marco Wallenborn, O. Petters, Daniela Rudolf, H. Hantmann, Mandy Richter, Peter Ahnert, Leili Rohani, J.J. Smink, G.C. Bulwin, W. Krupp, Ronny M. Schulz & Heidrun Holland
European Cells and Materials Vol. 35, 225–241 ( 2018)
doi: 10.22203/eCM.v035a16 - Generation of human induced pluripotent stem cells using non-synthetic mRNA
L Rohani, C Fabian, H Holland, Y Naaldijk, R Dressel, H Löffler-Wirth, H Binder, A Arnold & A StolzingStem Cell Res, May;16(3), 662–672 (2016)
doi: 10.1016/j.scr.2016.03.008 - Preclinical good laboratory practice-compliant safety study to evaluate biodistribution and tumorigenicity of a cartilage advanced therapy medicinal product (ATMP)
Matthias Zscharnack, Christoph Krause, Gabriela Aust, Christian Thümmler, Frank Peinemann, Thomas Keller, Jeske J. Smink, Heidrun Holland, Jeremy S. Somerson, Jens Knauer, Ronny M. Schulz & Jörg Lehmann
Journal of Translational Medicine 13: 160, (2015)
doi: 10.1186/s12967-015-0517-x